来源:山东省兽医协会
一、 概念:
首先引用《高通量宏基因组测序技术检测病原微生物的临床应用规范化专家共识》对宏基因组测序的描述:
“宏基因组下一代测序(metagenomics next‑generation sequencing,mNGS)技术应是通过直接提取临床样本中的核酸进行文库构建和高通量测序,利用生物信息学算法分析样本中包含的病原微生物序列的种类及耐药基因和毒力基因,能够快速并且准确地检测各种临床样本中的病原微生物。
利用mNGS技术进行病原微生物检测需经样本前处理、核酸提取、文库制备、上机测序并满足测试的质量控制要求后,采用特定算法软件与专用的病原微生物数据库进行比对,实现对病毒、细菌、真菌、寄生虫及非经典微生物等的检测。mNGS 技术不依赖培养,对常见病原微生物检验阴性、经验治疗失败、不明原因的危急重感染的病原学诊断以及新发突发传染病的病原体发现具有独特价值。”
对于宏基因组测序的概念在不同领域还有一些差异,医学领域指的是包含所有病毒、细菌、真菌、寄生虫等微生物。但是在一些科研测序领域,宏基因组测序只包含DNA的生物,宏转录组测序测RNA的微生物。对于动物疫病的宏基因组测序一定是包含所有DNA和RNA病毒、细菌、真菌、寄生虫等微生物的测序。在临床检测中只检测DNA或者一个样品一部分做DNA测序,另一部分做RNA测序都是无法满足临床需求的。

二、高通量测序技术在动物疫病检测的应用方向
1.按用途
世界动物卫生组织(World Organisation for Animal Health,WOAH)的陆生动物诊断疫苗手册中有对高通量测序技术的要求。
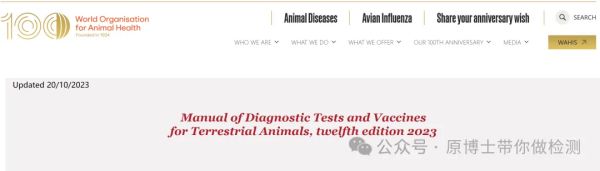

其中对于动物疫病的应用方向列了10条。按照应用场景主要分为检测、流调和特定病原研究几个方向。我重点关注多病原检测、未知病原检测、疫病流行病学调查和病原的溯源几个方面
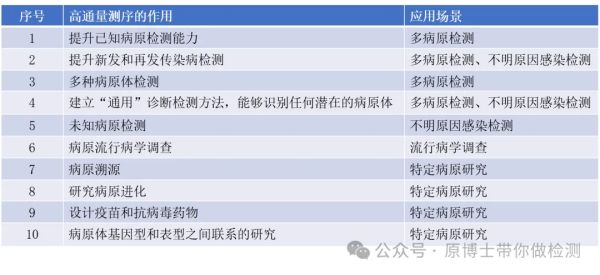
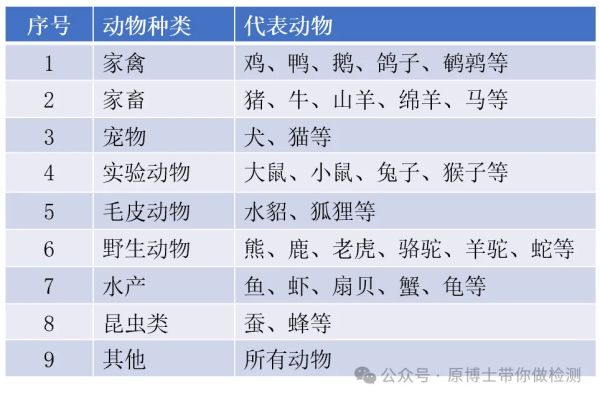
2.按动物种类
3.按病原种类
理论上包含所有与动物相关的微生物(含人的病原体):病毒、细菌、真菌、寄生虫等。

三、动物疫病宏基因组测序基本流程
宏基因组测序通常分为湿实验和干实验。
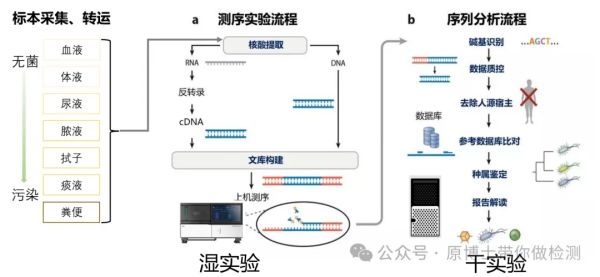
(图片来自网络,侵删)
1.湿实验
湿实验流程是指对待测标本进行前处理、核酸提取、文库构建、高通量测序,以获得核酸测序数据的实验室操作环节。
1.1样品采集和处理
在医学领域,样品的采集通常在医院完成,样品类型相对固定,采样标准化和规范化,采样过程也可以根据需要进行无菌操作。在兽医领域样品可包含你能想象的所有样品类型。活体采样可以有鼻拭子、唾液、全血、血浆、粪便、奶样、流产物等。如果对死亡动物进行尸体剖检,则需要专业的兽医结合临床症状与病理变化对病变组织进行采样,样品类型可包含所有脏器。对于不同的样品类型还需要采用不同的前处理方式,以保证组织中病原微生物能够有效释放。
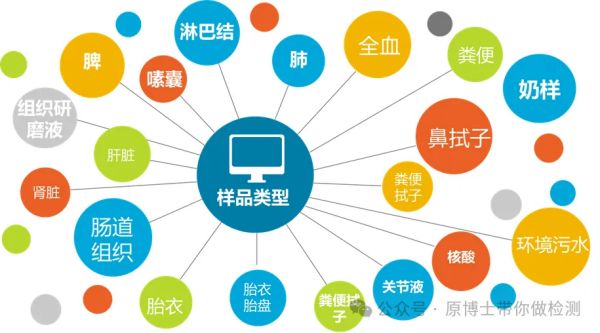
样品类型
1.2 核酸提取
由于样品类型繁多复杂,结合不同样品的前处理方式,需要采用比常规荧光定量PCR使用更高效的核酸提取试剂,以保证提取核酸的产量和质量。对于组织样品还需要进行去宿主基因组来降低背景干扰。
1.3 文库构建
动物病原宏基因测序需要采用DNA和RNA共建库的策略。实验室在选择方法前需对核心技术环节(如标签、接头的连接)质量进行评估,主要质量指标包括单/双标签测序,连接效率等。此外还应考察建库前核酸投入量的下限和上限,确保取得的文库在核酸插入片段的大小分布上满足所使用的高通量测序仪、测序模式和读长,以及后续生信分析对片段大小的要求,以避免过度片段化或片段化不足的情况。
1.4 高通量测序
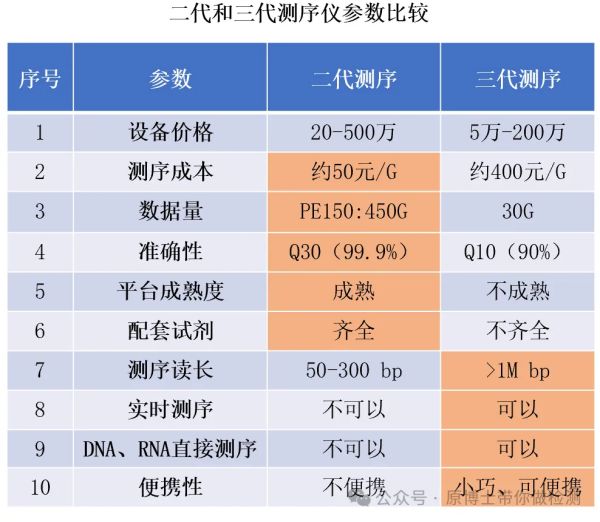
二代测序以illumina和华大智造为代表,三代测序以ONT、pacbio和齐碳生物为代表,二代三代的叫法只是技术产生的早晚,并不代表技术先进性。测序平台的选择建议从设备价格、测序成本、数据量、准确性、平台成熟度、配套试剂、测序读长、实时测序、DNA、RNA直接测序、便携性等十个方面综合评价。通常来说,二代测序具有准确性高、测序通量高和单位成本低、平台成熟度高、配套试剂全的优势;三代测序有仪器便宜、测序读长长、可以实时测序、RNA直接测序、仪器小巧便携的优势。每种技术都有自己的优缺点,选择适合自己的才是最好的。
2.干实验
干实验流程是指对测序数据进行生物信息学分析及结果审核和报告的过程。
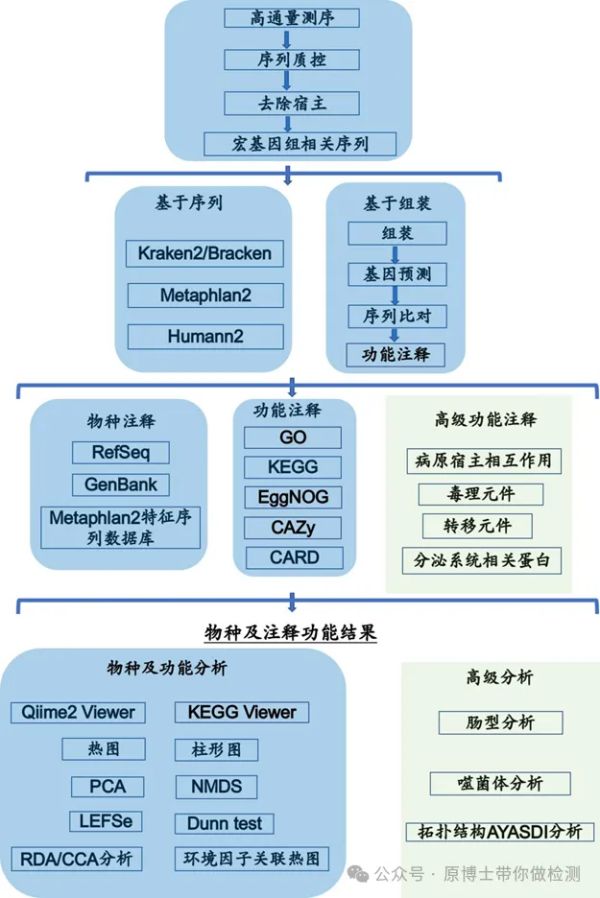
mNGS分析流程图
中科基因动物病原微生物信息分析系统
2.1 下机数据标签识别和序列拆分
高通量测序文库常批量混合上机(多份样本混合后加入一张芯片),在测序后需通过识别序列上的标签(称为barcode,一般6~10 bp,属人工合成的已知序列)区分不同样本。
2.2 数据质控
下机数据经拆分后即得到每个样本的测序数据,需要进行数据质量过滤,包括过滤测序接头、低质量序列、低复杂度序列、重复序列等,将获得的高质量读长序列作为微生物鉴定的输入数据。所谓低质量序列指未达到以下指标:Q30碱基数量占比>80%、接头污染比例不超过1%、有效序列长度不小于50 bp、数据的有效比对率应大于70%的基本特征。过滤软件有SOAPnuke、Trimmomatic、fastp及Adapter Removal等。
2.3 过滤宿主序列
人的宏基因组测序只需要去除人的基因组,对于动物的宏基因组测序,需要去除每种动物的宿主基因组,否则会降低测序的灵敏度并引入假阳性的结果。中科基因动物病原微生物信息分析系统中内置了下表中的宿主数据库,并且可以根据客户的需要随时进行补充。
中科基因目前已经开展了对猪、牛、羊、鸡、鸭、鹅、鸽、犬、猫、兔、大鼠、小鼠、骆驼、狐狸、老虎、细胞培养物、鲑鱼、污水等物种的病原宏基因组测序,积累了大量数据和丰富的经验。

2.4 物种注释
物种注释是病原宏基因组检测最核心的内容之一,主要是将通过质量控制的非宿主序列与微生物参考数据库比对,或者经过从头组装成contigs/scaffolds后再比对到微生物参考数据库。常用的分析流程有:基于Reads(有参)的宏基因组分析流程、基于组装(De novo)的宏基因组分析流程和宏基因组分箱分析流程。常用的物种分析软件有CLARK、Kraken2、LMAT、MEGAN、MetaPhlan、HUMAnN、QIIME、mOTU及MetaPhyler等。中科基因采用大量临床测序数据对宏基因组分析流程进行了大量的测试和参数优化,目前的分析流程更适合动物病原的数据分析。
2.5 参考数据库
微生物参考数据库的选择显著影响物种注释分类的结果 。目前没有任何一个公共数据库能够包含所有的潜在病原体的基因组信息(假阴性风险),且数据库中不可避免地存在一些注释错误或污染的序列(假阳性风险)。同时开展动物病原宏基因组测序的实验室需要不断对动物病原数据库进行更新,以应对临床上病原的变异。
中科基因动物病原微生物信息分析系统内置了专门针对不同动物病原的数据库,可以更精准的识别不同动物的致病病原并定期进行最新流行毒株的病原更新。

中科基因病原数据库
2.6 报告
在mNGS 报告中应含总序列数、微生物序列数、物种信息、测序覆盖度、及相对丰度。
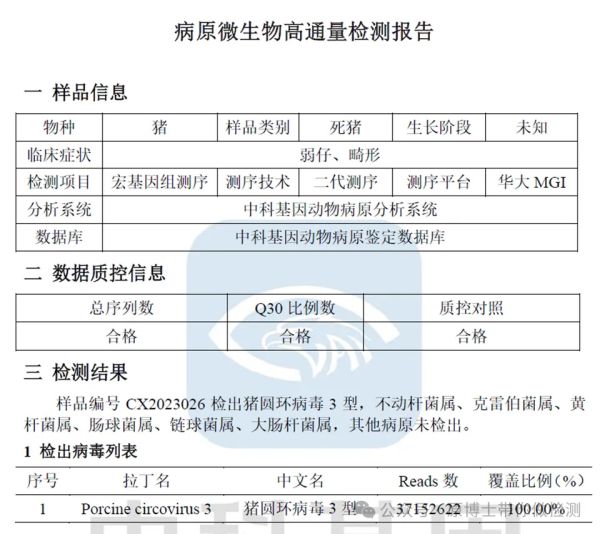
中科基因mNGS测序报告
中科基因动物病原微生物信息分析系统将识别的病原自动匹配中文注释,注释信息包含:物种中英文名称、引起的疾病名称、临床症状、易感动物、传播途径和病原在世界动物卫生组织的分类,我国农业农村部一二三疫病中的分类,以及是否为人畜共患病。还可对病毒进行序列提取和拼接,计算测序覆盖度和深度。
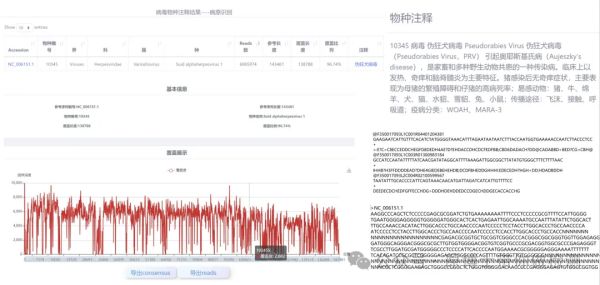
中科基因mNGS测序结果及注释
2.7 深度分析
部分客户对于宏基因组测序获得的基本信息需要更深一步的分析,中科基因可根据客户需要进行定制化分析:包括物种分类、进化树、堆叠图、热图、网络图等。所绘制的图都为了描述动物病原的组成、分类和进化等特征,而非很多华而不实,无法得出任何结论的分析。
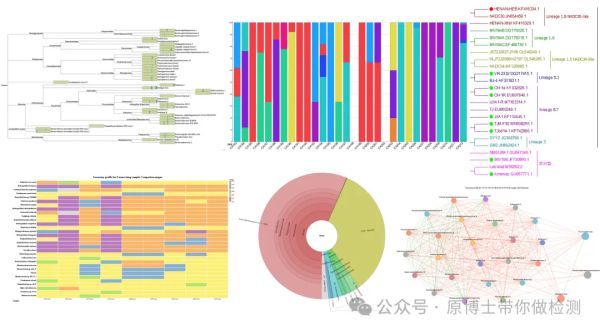
中科基因mNGS测序深度分析图
四、动物疫病宏基因组测序面临的挑战
1. 技术成本高
目前mNGS 的技术成本仍然较高,这限制了其在动物疫病领域的广泛应用。
2. 数据分析门槛高
mNGS 产生的数据量巨大,需要专用服务器和专业的生物信息学分析人员进行分析,这对于一些基层动物疫病防控机构来说存在困难。
3. 标准化体系不完善
mNGS 在动物疫病领域的应用包括数据库、分析工具、判定标准方面尚缺乏标准化体系,这影响了检测结果的准确性和可比性。
4.技术推广和培训:
mNGS目前在动物疫病检测领域应用较少 ,需要培训科研人员和从业者认识该技术的应用场景,以便使mNGS能够应用于临床的诊断。
随着技术的进步和成本的降低,mNGS 将在动物疫病领域得到越来越广泛的应用。mNGS 将有助于提高动物疫病的诊断效率、监测病原体变异、指导疫苗研发和疫情防控,为动物疫病防控提供新的技术手段。

中科基因动物病原测序业务
联系人:崔先生
联系电话:18853858069
地址:北京市大兴区中关村科技园大兴生物医药产业基地永大路38 号